M13mp18 RF I DNA 收藏
Download:
- isoschizomers |
- compatible ends |
- single letter code
特性
• 蓝/白斑筛选
浓度
100 μg/ml
分子量/长度
7,249 bp

100 μg/ml
7,249 bp
500 μg/ml。







NEBuffer 1.1: 25%
NEBuffer 2.1: 25%
NEBuffer 3.1: 25%
CutSmart Buffer: 100%
CutSmart、重组酶、省时酶。
CutSmart 缓冲液,37℃。
热失活:65℃ 20 分钟。
5,000 units/ml。
对 dam、dcm 和哺乳动物 CpG 甲基化均不敏感。
B s p 1 2 8 6 l 识别六种不同的序列:GGGCCC、GAGCCC(互补GGGCTC)、GTGCCC(互补 GGGCAC)、GAGCAC、GTGCAC 和 GAGCTC(互补 GTGCTC)。
如下条件可能出现星号活性:延时酶切、高酶浓度或甘油浓度 > 5%。

产品于 2020年 1 月 2 日停产.
67 kDa。
10,000 units/ml。
Monarch Bead Retainers are plastic parts, resembling spin columns without a membrane, designed and used to retain the Monarch DNA Capture Beads (NEB #T3005) during the Monarch HMW DNA extraction workflow. They are compatible with Monarch Collection Tubes II (NEB #T2018).
Storage Temperature
25°C





几分钟之内将抗体及融合蛋白完全去糖基化

75℃ 10 分钟。
无糖苷外切酶污染,无糖苷内切酶F1、F2 和 F3 活性,无蛋白水解活性。



T4 Rnl2tr KQ 是 T4 RNA 连接酶 2,截短型(NEB#M0242)的双点突变体。 K227 的突变降低了赖氨酰腺苷化。由于降低了 T4 Rnl2tr 从接头将腺苷基团转移到 RNA 5´ 磷酸基的活性, K227Q 可以减少 T4 Rnl2tr 非特异性的连接(串联体和环)。在 T4 Rnl2tr K227Q 中进一步突变 R55K,提高了酶的连接活性,使其与野生型 T4 Rnl2tr 连接活性水平一致。
因为不再使用 ATP,而改用预腺苷化接头,同时降低赖氨酰腺苷化酶的活性,所以连接反应的背景可以降至最低。该酶已用于二代测序 Small RNA的文库构建中,优化了接头的连接过程。
重组 E. coli 菌株,携带有 MBP 融合表达的质粒,该质粒克隆有含编码 T4 RNA 连接酶 2 的前249 个氨基酸序列。 T4 Rnl2tr K227Q 是将 227 位的赖氨酸突变为谷氨酸。 T4 Rnl2tr KQ 分别在 55 和 位置进行了突变,精氨酸突变为赖氨酸、赖氨酸突变为谷氨酰胺。
10X T4 RNA 连接酶反应缓冲液
50% PEG 8000
无单链和双链 DNA 核酸内切酶、外切酶、 RNase 以及磷酸酶的污染。
200 单位指 10 μl 反应体系中,25℃ 条件下,1 小时能将 80% 的 31-mer RNA 连接到预腺苷化 17-mer DNA 末端 [miRNA 通用克隆接头(NEB #S1315)] 所需的酶量。单位活性检测条件请登陆 www.neb-china.com 或 www.neb.com。
200,000 units/ml。
有关该产品特性和应用的参考文献请登陆 www.neb-china.com,www.neb.com。










NEBuffer 1.1: <10%
NEBuffer 2.1: 50%
NEBuffer 3.1: 10%
CutSmart Buffer: 100%
CutSmart、重组酶、基因工程改造酶、省时酶、高保真酶。
CutSmart 缓冲液,37℃。
20,000 units/ml。
对哺乳动物基因组 DNA CpG 甲基化敏感。




40,000 units/ml。









NEBuffer 1.1: <10%
NEBuffer 2.1: <10%
NEBuffer 3.1: <10%
CutSmart Buffer: 100%
CutSmart、重组酶、省时酶。
CutSmart 缓冲液,37℃。
10,000 units/ml。
对哺乳动物基因组 DNA CpG 甲基化敏感。
Fnu4Hl 产生的 DNA 片段包含有单碱基的 5´ 突出端,比平末端更难连接。











CutSmart 缓冲液,37℃。
浓度:20,000 units/ml。
对 dcm 和哺乳动物基因组 DNA CpG 甲基化敏感
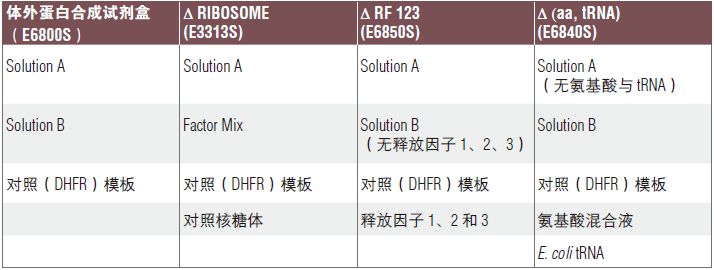
PURExpress 试剂盒采用 PURE 系统技术,该技术最早由东京大学 Takuya Ueda 博士发明,Biocomber(日本,东京)商业化为 PUResYsTeM®。
PURExpress® 体外蛋白合成试剂盒是新型无细胞转录/翻译系统,用于基因快速表达分析,是由大肠杆菌翻译所必需组份纯化而构成。PURExpress 系统无核酸酶和蛋白酶的污染,保护了模板 DNA 和 RNA,避免了蛋白的修饰及降解。转录和翻译过程仅需将两管试剂混合,一步反应即可完成,几个小时就可观察实验结果,PURExpress 系统大大地节省了宝贵的实验时间,特别适合于高通量技术。
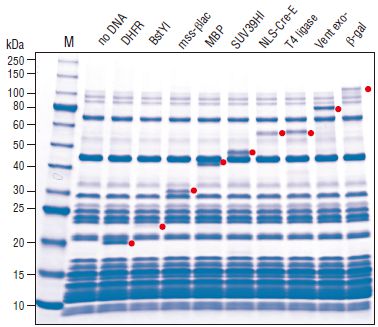
使用 PURexpress 体外蛋白合成试剂盒表达蛋白质。25 μl 反应体系中加入 250 ng 模板 DNA 和 20 单位 RNase 抑制剂,37℃ 反应 2 小时。各取 2.5 μl 反应液,进行 10-20% Tris-甘氨酸 SDS-PAGE 电泳分析。红点处指示的为目的蛋白。Marker M 是蛋白质分子量标准(NEB #P7703)
该专利产品的成分为蛋白质与缓冲液,当 PURExpress 系统合成或 E. coli S30 提取的目的蛋白具有较多二硫键时,能帮助其正确折叠。在合成反应开始时加入 PURExpress 二硫键增强剂,能够协助半胱氨酸硫醇的氧化,并纠正硫醇的错误氧化,从而提高可溶性蛋白及具有功能活性蛋白的产量。
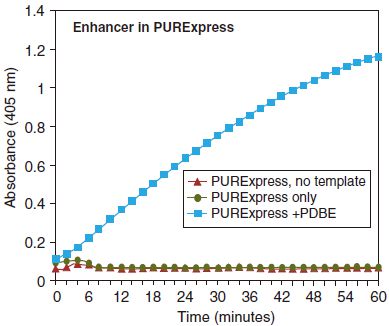
PURexpress 二硫键增强剂。(PDBE)促进活性 vtPA 的正确折叠。反应按照 PURExpress 系统说明进行,以 vtPA DNA 为模板。37℃ 下温育 2 小时后,取 5 μl 反应液进行活性检测,在 1 小时内监测发光底物的剪切反应。
此试剂盒中移除了核糖体,使得研究蛋白翻译的用户能够使用自己的修饰核糖体。试剂盒还提供单独一管的对照核糖体(2 次反应用量)。
释放因子通过识别 mRNA 序列上的终止密码子参与蛋白翻译的终止过程。采用 PURExpress 进行核糖体展示实验时,缺乏释放因子能够稳定 mRNA-核糖体-新生肽链形成的三元复合体。因此 cDNA 的回收率会更高。该试剂盒单独提供三种释放因子,用户能够自行选择在有或无释放因子的条件下进行蛋白质体外合成或核糖体展示。
该试剂盒单独提供 tRNA 与氨基酸,用户能够向反应体系中加入修饰过的氨基酸与 tRNA 混合液进行蛋白质合成反应。
大肠杆菌的 70S 核糖体由一个小亚基(30S)与一个大亚基(50S)组成。该核糖体试剂在 NEB 的 PURExpress体外蛋白合成试剂盒(NEB #E6800)中活性很高,在核糖体结构与功能研究中,能够用于药物筛选的靶标以及分离天然核糖体 RNA(5S、16S、23S)的起始材料。该产品溶液浓度为 33.3 mg/ml。
SNAP-Cell 阻断剂
CLIP-Cell 阻断剂
SNA-Surface 阻断剂









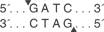
NEBuffer 1.1: 75%
NEBuffer 2.1: 100%
NEBuffer 3.1: 100%
CutSmart Buffer: 100%
CutSmart、重组酶、省时酶。
CutSmart 缓冲液,37℃。
热失活:65℃ 20 分钟。
5,000 和 25,000 units/ml。
对 dam 甲基化敏感,而完全同裂酶 Sau3Al 对 dam 不敏感。对哺乳动物基因组 DNA CpG 甲基化敏感。
BfuCl、Dpnll 和 Sau3Al 是 Mbol 的完全同裂酶。Mbol 产生一个 5´ GATC 突出端,能有效地与 BamHl、Bcll、Bglll、BstYl、Dpnll 或 Sau3Al 切割的片段相连接。
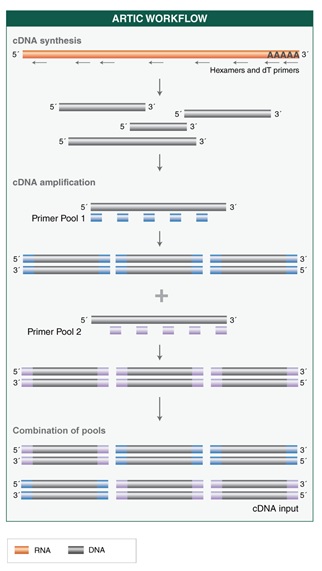
NEBNext® ARTIC SARS-CoV-2 文库制备试剂盒(适用于 Illumina 平台)
目前对 SARS-CoV-2 测序的可靠性和准确性的要求日益增长,为了满足上述需求,NEB 推出 NEBNext® ARTIC 文库制备试剂盒。这些试剂盒根据 ARTIC Network(1)原始流程开发,采用多重扩增子的方法对病毒进行全基因组测序。
SARS-CoV-2 RT-PCR 扩增性能优越。起始样本是 100 ng 人类通用参考 RNA(ThermoFisher QS0639)中的 10 – 10,000 份 SARS-CoV-2 病毒 gRNA 拷贝(ATCC VR-1986 和 VR-1991)。使用 NEBNext 已平衡的 ARTIC SARS-CoV-2 引物池对起始样本进行多重扩增子扩增。使用 Qubit™ dsDNA BR Assay Kit(ThermoFisher Q32850)测定平均扩增子产量。
在 Illumina 平台使用 NEBNext ARTIC SARS-CoV-2 引物池,可提高基因组覆盖度。基因组数据可视化图(IGV)显示了 SARS-CoV-2 基因组的覆盖度。起始样本是 100 ng 人类通用参考 RNA(ThermoFisher QS0639)中的 1,000 份 SARS-CoV-2 病毒 gRNA 拷贝(ATCC VR-1986 和 VR-1991)。分别使用两种引物对起始样本进行多重扩增子扩增:IDT ARTIC nCoV-2019 V3 Panel(“Standard”)和 NEBNext 已平衡的 ARTIC SARS-CoV-2 引物池。建库试剂为 NEBNext ARTIC SARS-CoV-2 FS 文库制备试剂盒(适用于 Illumina 平台)。使用 MiSeq® (2 x 75 bp)测序。测定每个碱基的覆盖深度,向下抽样(seqtk)Reads 数,使用 Bowtie 2 比对至 SARS-CoV-2 参考基因组(NCBI, NC_045512)。
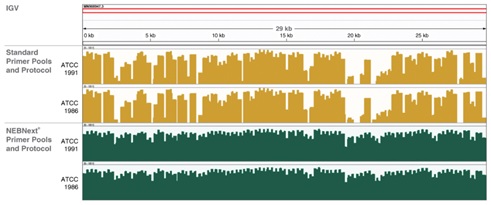
在 Oxford Nanopore Technologies 平台使用 NEBNext ARTIC SARS-CoV-2 引物池,完全覆盖基因组所需的 Reads 数更少。IGV 图显示了 SARS-CoV-2 基因组的覆盖度。使用 IDT ARTIC nCoV-2019 V3 Panel(“Standard”)或 NEBNext 已平衡的 ARTIC SARS-CoV-2 引物池,100 ng 人类通用参考 RNA(ThermoFisher QS0639)中的 1000 份 SARS-CoV-2 病毒 gRNA 拷贝(ATCC VR-1986 和 VR-1991)作为起始样本制备扩增子。使用 NEBNext ARTIC SARS-CoV-2 配套试剂盒(Oxford Nanopore Technologies)、Oxford Nanopore Technologies Native Barcoding Expansion kits 1-12(EXP-NBD104)、1 3-24 (EXP-NBD114)、Ligation Sequencing Kit(EXP-NBD114)、Ligation Sequencing Kit(SQK-LSK109)和 SFB Expansion Kit(EXP-SFB001)制备文库。使用 R9.4.1 flow cells 在 GridION 仪器上进行测序,并使用 Minimap2 比对数据。
NEBNext ARTIC SARS-CoV-2 RT-PCR 模块流程








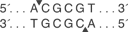
NEBuffer 1.1: 10%
NEBuffer 2.1: 50%
NEBuffer 3.1: 100%
CutSmart Buffer: 25%
重组酶、省时酶。
NEBuffer 3.1,37℃。
热失活:80℃ 20 分钟。
10,000 units/ml。
对哺乳动物基因组 DNA CpG 甲基化敏感。
1 μm 无孔超顺磁微粒(#S1420)或 2 μm 无孔超顺磁微粒(#S1421)。
关于本产品特点与应用的参考文献请登陆 www.neb-china.com,www.neb.com。
T1 噬菌体(fhuA2)、链霉素
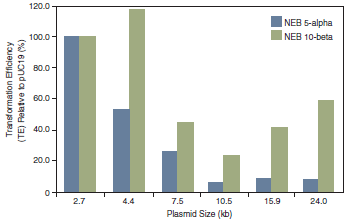





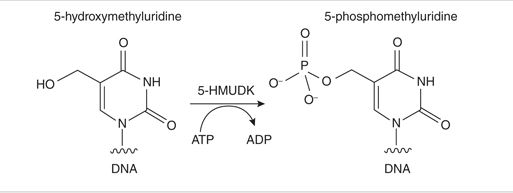
该酶是创新酶(Enzyme for Innovation, EFI)。创新酶工程由 NEB 发起,旨在为科研界提供独一无二的酶,从而为新的创新应用的发现创造条件。完整创新酶列表请登陆 www.neb.com/EnzymesforInnovation。
5-羟甲基尿苷 DNA 激酶(5-HMUDK)可将 ATP 的 γ-位磷酸基团转移到 DNA 链上的 5-羟甲基尿苷的羟甲基上
1X T4 DNA 连接酶反应缓冲液,37℃ 温育。
1 单位指在 20 μl 反应体系中,37℃ 条件下,30 分钟能保护 1 μg 枯草芽孢杆菌噬菌体 SP8 基因组 DNA 不被 NcoI-HF 限制性内切酶切割所需要的酶量。
20,000 units/ml
T1 噬菌体(fhuA2)







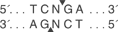
NEBuffer 1.1: 25%
NEBuffer 2.1: 100%
NEBuffer 3.1: 50%
CutSmart Buffer: 100%
CutSmart、重组酶。
CutSmart 缓冲液,37℃。
热失活:65℃ 20 分钟。
10,000 units/ml。
对 dam 甲基化敏感。
Hpy188l 产生的 DNA 片段包含有单碱基的 3´ 突出端,比平末端更难连接。
如下条件可能出现星号活性:延时酶切、高酶浓度或甘油浓度 > 5%。







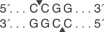
NEBuffer 1.1: 75%
NEBuffer 2.1: 100%
NEBuffer 3.1: 50%
CutSmart Buffer: 100%
CutSmart、重组酶、省时酶。
CutSmart 缓冲液,37℃。
20,000 和 100,000 units/ml。
对 dam、dcm 和哺乳动物 CpG 甲基化均不敏感。
Mspl 是 Hpall 的完全同裂酶。
NEBNext 酶学转化法甲基化建库相关产品
NEBNext 酶学转化法甲基化建库试剂盒
NEBNext 甲基化建库酶学转化法模块
NEBNext Q5U™ 预混液
NEBNext 酶学转化法甲基化建库多样本接头引物(双端检索引物)
虽然重亚硫酸盐测序是研究 DNA 甲基化的金标准,但这种转化方式会对DNA 造成损伤,导致 DNA 断裂、丢失和 GC 偏嗜。NEBNext 酶学转化法甲基化建库试剂盒 (EM-seq™) 提供了一种酶学方法代替全基因组重亚硫酸盐处理 DNA (WGBS) 的方法,并结合高效流程化的建库步骤,适用于 Illumina 平台测序。高效的 EM-Seq 酶促转化可最大程度地减少对DNA的损伤,结合提供的 NEBNext Ultra II 文库制备流程,最终产生的高质量文库可以从有限的测序数据中更灵敏的检测到 5-mC 和 5-hmC。
更卓越的 5-mC 和 5-hmC 检测灵敏度
更高的比对率
更均一的 GC 覆盖度
能从有限的测序数据检测到更多的 CpG 位点
更均一的二核苷酸分布
更长的文库插入片段
更高效的建库流程
提供转化模块


EM-Seq 能得到更高的文库产量。采用Covaris S2 仪器将 10 ng、50 ng 和 200 ng 不同起始量的人 NA12878 基因组 DNA 打断至 300 bp,同时作为 EM-Seq 和 WGBS 建库的起始样本。对于 WGBS 方法,使用 NEBNext Ultra II DNA 进行建库,随后采用 Zymo Research EZ DNA Methylation-Gold 试剂盒进行重亚硫酸盐转化。上述所有起始量,EM-Seq 都能使用更少的 PCR 循环得到更高的文库产量,表明 EM-Seq 显著减少了 WGBS 方法中的 DNA 损失。误差条表示标准差。

NEBNext 酶学转化法甲基化文库可获得更长的插入片段 。采用Covaris® S2 仪器将 50 ng 人 NA12878 基因组 DNA 打断至 300 bp,同时作为 EM-Seq 和 WGBS 建库的起始样本。对于 WGBS 方法,使用 NEBNext Ultra II DNA 进行建库,随后采用 Zymo Research EZ DNA Methylation-Gold™ 试剂盒进行重亚硫酸盐转化。两个文库均使用 Illumina MiSeq (2 X 76 bases) 测序,片段大小采用 Picard 2.18.14 测定。图中绘制了每个插入片段出现的频率标准化后的数据,结果如图:EM-Seq 建库后的插入片段比 WGBS 方法得到的插入片段更长,说明:酶学转化法不会对 DNA 造成损伤,而重亚硫酸盐处理的 DNA 有严重损伤。

相较于 WGBS,EM-Seq 在更低的测序深度情况下能检测到更多的 CpG 位点,并能实现更卓越的 GC 均一覆盖度。采用Covaris S2 仪器将 10 ng, 50 ng, 200 ng 不同起始量的人 NA12878 基因组 DNA 打断至 300 bp,同时作为 EM-Seq 和 WGBS 建库的起始样本。对于 WGBS 方法,使用 NEBNext Ultra II DNA 进行建库,随后采用 Zymo Research EZ DNA Methylation-Gold™ 试剂盒进行重亚硫酸盐转化。两个文库均使用 Illumina NovaSeq® 6000 (2 X 100 bases) 测序,使用 bwa-meth 0.2.2 将 测序数据 与 hg38 进行比对。
A: CpG 位点检测:通过分析 3.24 亿双端数据得到 EM-Seq 和 WGBS 文库的 CpG 位点覆盖度,其中每条链都独立计数,最终得到最多 5600 万个可能的 CpG 位点。结果显示:EM-Seq 在更低测序深度能检测到更多的 CpG 位点。
B: GC 覆盖度:使用 Picard 2.17.2 计算 GC 覆盖度,图中显示不同 GC 含量时 (0-100%),标准化后覆盖度的分布情况。结果显示:EM-Seq 文库显著提高 GC 覆盖度的均一性,无 AT 过度测序,也无 GC 测序不足,后两项都是 WGBS 文库的典型缺陷。

相较于 WGBS,EM-Seq 在更低的测序深度情况下能检测到更多的 CpG 位点。采用 Covaris S2 仪器将 10 ng、50 ng 和 200 ng 不同起始量的人 NA12878 基因组 DNA 打断至 300 bp,同时作为 EM-Seq 和 WGBS 建库的起始样本。对于 WGBS 方法,使用 NEBNext Ultra II DNA 进行建库,随后采用 Zymo Research EZ DNA Methylation-Gold™ 试剂盒进行重亚硫酸盐转化。两个文库均使用 Illumina NovaSeq® 6000(2 X 100 bases)测序,使用 bwa-meth 0.2.2 将测序数据与 hg38 进行比对。通过分析 3.24 亿双端数据得到 EM-Seq 和 WGBS 文库的 CpG 位点覆盖度。
图中显示了 EM-Seq 和 WGBS 两种方法在不同起始量,至少 1X 和 8X 测序深度下检测到的独有和共有的 CpG 位点。EM-Seq 在至少 1X 测序深度下比 WGBS 多检测到 20% 以上的 CpG 位点。而在至少 8X 测序深度下,CpG 位点覆盖度差异增加至 2 倍。
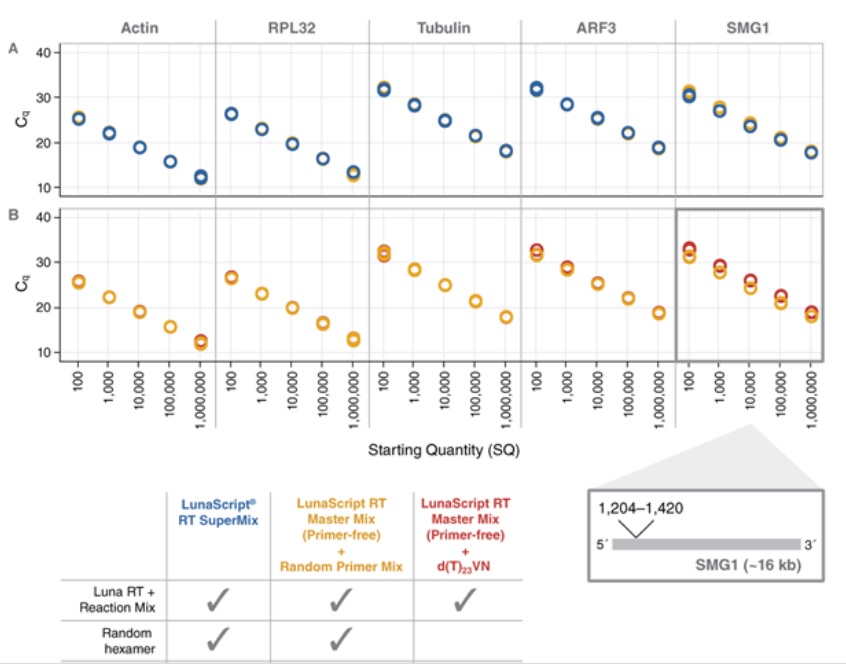
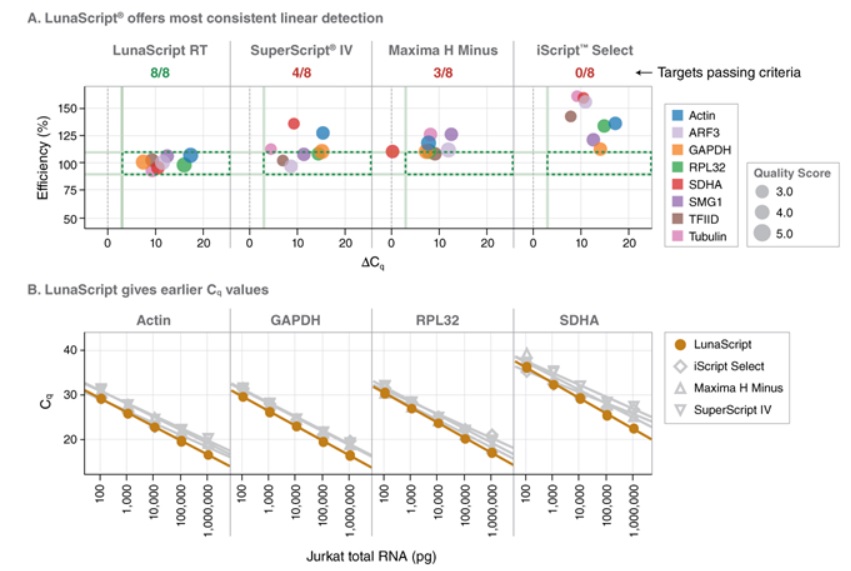
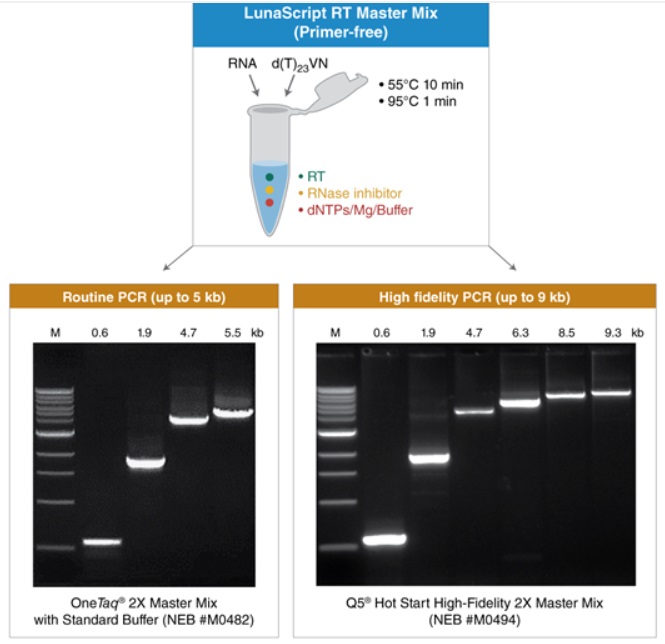
使用 LunaScript 反转录预混液试剂盒(无引物)将 1 μg Jurkat 总 RNA 反转录成 cDNA,引物为 d(T))23VN,使用标准反应条件 55℃ 孵育 10 分钟,95℃ 孵育 1 分钟。之后使用不同 PCR 扩增试剂对 cDNA 产物进行检测。对于 5 kb以内的常规应用,建议使用 OneTaq 2X 预混液(NEB #M0482);对于高保真扩增,建议使用 Q5 热启动超保真 2X 预混液(NEB #M0494);对于长片段高产量扩增,建议使用 LongAmp Taq 2X 预混液(NEB #M0287)(数据未显示)








NEBuffer 1.1: 25%
NEBuffer 2.1: 25%
NEBuffer 3.1: <10%
CutSmart Buffer: 100%
CutSmart、重组酶。
CutSmart 缓冲液,37℃。
10,000 units/ml。
对哺乳动物基因组 DNA CpG 甲基化敏感。
由于 Nael 具有底物位点优势效应(有关底物位点优势效应详情请登陆:www.neb-china ,.comwww.neb.com),所以切割载体 pBR322 DNA 速度缓慢,而 Nael 的同裂酶 NgoMⅣ 的底物位点优势较弱。


PNGase F
PNGase F ( 无甘油 )
PNGase F, 重组酶
PNGase F (无甘油), 重组酶
RNase B(对照底物)
Endoglycosidase Reaction Buffer Pack

36,000 daltons。
500,000 units/ml。
停产通知:该产品将于 2021 年 12 月 15 日停产,推荐替代产品为 #E7546S
The NEBNext® Ultra™ End Repair/dA-Tailing Module is optimized to convert 5 ng–1 μg of fragmented DNA to repaired DNA having 5′ phosphorylated, 3’ dA-tailed ends. The module is optimized for use with
NEBNext Ultra Ligation Module (NEB #E7445), and is part of the original Ultra workflow, which enables
library preparation from 5 ng – 1 μg of input DNA in a streamlined protocol.
NEBNext End Prep Enzyme Mix
NEBNext End Repair Reaction Buffer
-20°C
请登陆 NEBNextSelector.neb.com 可以使用在线产品选择工具进行产品选择。









NEBuffer 1.1: 75%
NEBuffer 2.1: 100%
NEBuffer 3.1: 10%
CutSmart Buffer: 100%
CutSmart、重组酶、省时酶。
CutSmart 缓冲液,65℃。
20,000 units/ml。
10%。
对哺乳动物基因组 DNA CpG 甲基化敏感。
BstBl 是 Fspll 的完全同裂酶。